L’ADN, un langage comme un autre ?

Comparable à une langue, l’ADN porte et transmet des messages exprimés par nos gènes. Ces informations génétiques, massives et codées, sont déchiffrées et lues par les scientifiques grâce à l’informatique. Mathieu Bergé, biologiste, et Roland Barriot, bio-informaticien au Centre de biologie intégrative jouent les traducteurs. Étude de cas d’une bactérie bavarde aux méthodes de communication singulières : le pneumocoque.
Par Clara Mauler, journaliste
L’ADN : un alphabet à 4 lettres
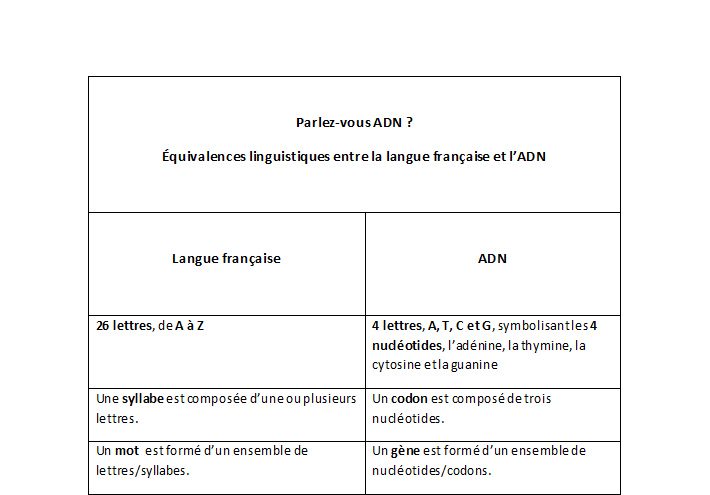
Si la langue française utilise 26 lettres et le langage informatique recourt aux 0 et 1 pour transmettre des informations, en biologie, ce sont 4 lettres qui codent toute l’information génétique comprise dans notre ADN (acide désoxyribonucléique). « Conceptuellement l’ADN peut être vu comme un langage », résume Mathieu Bergé, enseignant-chercheur de l'Université Toulouse III - Paul Sabatier.
En effet, la molécule d’ADN est constituée d’un enchaînement de quatre types de petites molécules, appelées les nucléotides et symbolisées par les lettres A, T, C et G pour adénine, thymine, cytosine et guanine.
Les gènes : des mots à milliers de caractères
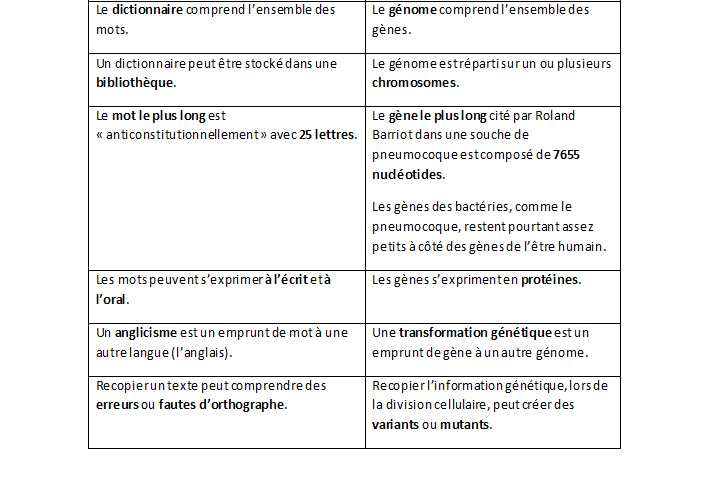
Notre alphabet forme des syllabes qui composent des mots écrits dans un dictionnaire. Les nucléotides s’assemblent en codons qui composent des gènes inscrits sur la molécule d’ADN. « C’est un alphabet à quatre lettres pour former des mots à milliers de caractères », illustre Roland Barriot. Si le mot le plus long de la langue française atteint les 25 lettres avec « anticonstitutionnellement », les gènes peuvent contenir une suite de lettres bien plus importante. « À la fin des années 1990, il a été mis en évidence que le pneumocoque est composé de 2 millions de caractères. Ses 2000 gènes approchent chacun les 1000 caractères », expliquent les chercheurs.
Au début des années 2000, le génome humain est « lu » ou séquencé. Ses 3 milliards de nucléotides sont connus, mais les gènes ne sont pas tous identifiés. Autrement dit, la succession des lettres est lisible, mais le découpage en mots n’est pas encore déterminé.
« C’était formidable de dire qu’on avait séquencé le génome humain, mais on ne savait pas lire le dictionnaire, on ne savait pas où commençaient et où finissaient les mots ! »
résume Roland Barriot, enseignant-chercheur de l'Université Toulouse III - Paul Sabatier.
Et à une lettre près, le sens et l’information que donne un mot changent. « Vous remplacez un « c » par un « d » et vous passez de « diplococus », le nom initialement donné au pneumocoque qui mesure quelques micromètres, au « diplodocus » long de 30 mètres ! », plaisante Mathieu Bergé.
Aujourd’hui, à moins d’une erreur de lecture, les gènes du génome humain semblent être tous identifiés. « Cela dépend de la fiabilité des méthodes de prédictions pour détecter la présence d’un gène. Certains gènes prédits peuvent ne pas en être, et inversement, on n’est pas certain de ne pas en avoir manqué quelques-uns », précise le bio-informaticien.
Exprimer des informations en protéines
Si la langue française s’exprime à l’écrit et à l’oral, l’information comprise dans les gènes s’exprime en protéines. L’opération s’effectue en deux étapes : la transcription et la traduction. Un gène est tout d’abord transcrit en ARN, une molécule intermédiaire, puis traduit en protéine. Chaque codon du gène correspond à un acide aminé, la base constituante des protéines. « Comme si on ne pouvait pas sortir un livre d’une bibliothèque, on ne peut pas sortir l’ADN du chromosome qui le porte, donc on recopie les passages du livre, lettre par lettre », image Mathieu Bergé.
L’informatique au service de la biologie
L’essor de la bio-informatique au milieu des années 2000 aide les scientifiques à analyser toute cette information codée. Au-delà de l’utilisation de méthodes existantes en informatique, mathématiques et statistiques, la bio-informatique conçoit ses propres méthodes de traitement de données adaptées aux questions biologiques. « Les méthodes développées sont devenues de plus en plus intégrées, voire essentielles, aux travaux de recherches menés en génétique et dans d’autres disciplines de la biologie », souligne Roland Barriot.
Un véritable changement d’échelle s’opère dans les stratégies de recherche en biologie. Quand une manipulation en laboratoire permettait d’observer quelques gènes, une simulation sur ordinateur en traite des milliers et offre une approche globale au niveau d’un organisme. « Il y a vingt ans, un chercheur pouvait avoir fait toute sa carrière sur l’étude de deux ou trois gènes chez un organisme », se souvient Mathieu Bergé. « On détruisait un gène soupçonné important dans une cellule et on observait in vivo ce qui se passait. Avec le passage à la bio-informatique, on s’est mis à étudier le comportement de tous les gènes simultanément dans une même cellule », complète-t-il.
Le pneumocoque, champion des anglicismes ou l’emprunt de gènes
Les équipes de Mathieu Bergé et Roland Barriot ont ainsi collaboré à l’étude de la bactérie du pneumocoque (Streptococcus pneumoniae). Ce micro-organisme unicellulaire, aux 2000 gènes, a la capacité de chercher et intégrer des gènes extérieurs, provenant d’autres bactéries, dans son propre génome. Il effectue cette opération en réaction par exemple à un stress chimique, comme la présence d’antibiotiques.
La langue française emprunte bien quelques mots à ses voisins européens pour pouvoir exprimer certaines informations. Des anglicismes se révèlent fort utiles dans certaines situations, si la faim vous amène à commander un « sandwich » ou si vous cherchez votre « smartphone » égaré.
Sur toute une population de pneumocoques qui effectuent cette « transformation génétique », certains individus vont récupérer des fragments d’ADN intéressants. « Le pneumocoque acquiert de nouveaux gènes qui lui donnent un avantage pour s’adapter à un nouvel environnement », révèle Mathieu Bergé. Subsistent alors les individus modifiés résistants à un stress, tel que les antibiotiques.
La capacité à effectuer cette « transformation génétique » est permise par l’expression de gènes précis, sous forme de protéines. L’objectif des scientifiques est de retrouver quels sont les gènes et protéines acteurs de la transformation. Une fois identifiés, ils peuvent devenir les cibles de traitements médicamenteux. Les acteurs mis hors-jeu, la transformation ne se ferait plus et l’adaptation au nouvel environnement serait nulle. Pour cela, un modèle bio-informatique simule une multitude de scenarii où l’absence d’un gène donné et ses conséquences au sein de la cellule sont observés.
Les cellules discutent entre elles
Le Centre de biologie intégrative développe une approche à plusieurs échelles : de la cellule isolée, à l’ensemble d’un organisme jusqu’aux interactions entre cellules, organismes, familles de bactéries… Cette dynamique de biologie intégrative et biologie des systèmes répond aux défis actuels en recherche biologique.
« Le modèle développé par le laboratoire pour l’étude du pneumocoque a été réalisé à l’échelle d’une cellule isolée. Mais il serait également intéressant d’observer les interactions des cellules entre elles »
précise Roland Barriot.
« Les cellules peuvent échanger du matériel génétique. Elles échangent aussi des molécules pour communiquer, partager des messages généraux du type « Je suis là ! » ou pour se dire « Moi je suis stressée ! ». Une cellule dit alors à la suivante « Transforme-toi aussi ! » »
explique Mathieu Bergé.
Les générations de cellules se passent le mot
La bio-informatique permet également de rechercher si d’autres familles de bactéries ont un mécanisme similaire à celui du pneumocoque et auraient une capacité à s’adapter à des stress.
Quand une cellule se divise, l’information génétique est recopiée. Mais il existe parfois des erreurs de copie qui créent des « mutations » ou « variants ». Une nouvelle famille apparaît avec des mécanismes en commun et certains différents. « Ces erreurs aléatoires se produisent assez fréquemment. C’est ce qu’on observe avec le coronavirus et ses variants actuellement », souligne le biologiste. Les virus utilisent en effet des cellules « hôtes » pour se répliquer et passer l’information génétique.
La phylogénétique retrace ces modifications génétiques et la genèse de nouvelles familles.
« On essaie de remonter l’arbre généalogique des bactéries, savoir qui a muté et ainsi déduire dans quelles autres espèces ce qui a été découvert dans une, reste valable dans une autre »
explique Roland Barriot.
Pour cela, la bio-informatique compare différents génomes et cherche à rapprocher des gènes se ressemblant. Des milliers de génomes de bactéries sont aujourd’hui connus et représentés informatiquement. Ils peuvent ainsi être lus et comparés entre eux. « On cherche à rapprocher des gènes ou « mots » qui se ressemblent sur différents génomes pour remonter le fil. Le « mot » qu’avait l’espèce ancestrale, chaque bactérie l’a aujourd’hui avec son accent, sa spécificité », détaille Roland Barriot.
Les bactéries, un livre ouvert ?
Des mots inscrits dans le génome du pneumocoque, aux discussions entre cellules jusqu’à la transmission d’informations entre générations de bactéries, la bio-informatique permettrait presque de lire l’ADN comme un livre ouvert.
Le parallèle entre ADN et langage atteint aujourd’hui son paroxysme. « D’autres équipes de recherche essaient de coder de l’information avec le codage ADN », révèle Mathieu Bergé. Des données et fichiers stockés informatiquement, codés en 0-1, seraient transformées en codage A-T-C-G pour passer du numérique au biologique. Une séquence de nucléotides contenant l’information serait fabriquée, placée dans une molécule autonome insérée dans une bactérie. Le stockage au sein de bactéries serait moins volumineux et plus durable que sur CD ou disque dur ! L’information se conserverait en maintenant la population de bactéries vivantes.
« On pourrait ainsi avoir une bibliothèque dans une bactérie ! »
conclut le biologiste.
Pour aller plus loin
- Dynamic Modeling of Streptococcus pneumoniae Competence Provides Regulatory Mechanistic Insights Into Its Tight Temporal Regulation, Weyder Mathias, Prudhomme Marc, Bergé Mathieu, Polard Patrice, Fichant Gwennaele, Frontiers in Microbiology, Volume 9, 2018
- L’ADN sera-t-il l’avenir du stockage de données ? Irène Tanneur, The Conversation, 24 mai 2021
CBI : Centre de biologie intégrative - Unité mixte de recherche de biologie moléculaire, cellulaire et du développement (CNRS, Université Toulouse III - Paul Sabatier)



